PICRUSt2预测群落功能-v2.6.2(2025年4月更新最新版本)
前言
PICRUSt2(Phylogenetic Investigation of Communities by Reconstruction of Unobserved States 2)是一种基于系统发育标记基因(如16S rRNA)预测微生物群落功能组成的生物信息学工具。它通过将测序获得的OTU/ASV序列与参考基因组数据库(如KEGG、COG、MetaCyc等)进行比对,利用预先计算的系统发育关系模型,推断微生物群落可能携带的功能基因、代谢通路及酶活性。相较于初代PICRUSt,PICRUSt2提高了预测准确性,支持更广泛的数据库和自定义参考数据,并整合了跨分类层级的功能注释。该工具广泛应用于生态学、医学及环境微生物学研究,但需注意其预测结果依赖于参考数据库的完整性,需结合实验进一步验证。
GitHub链接:PICRUSt2官网
1 准备工作:
需要至少16G内存的Linux环境下使用
如果没有安装Linux环境,参考:Windows 11 安装Ubuntu 20.04 LTS子系统 - leo朵
1.1 习惯性设置screen,事倍功半,保证和服务器断开连接也不中止任务
# 新建一个名为screen-name的会话窗口(screen-name可以自定义命名)
screen -S screen-name
# 断开名为screen-name的screen进程
# 使用前该screen进程的状态必须是Attached,使用后状态变为Detached
screen -d screen-name
# 返回到名为screen-name的会话窗口
screen -r screen-name
# 终止名为screen-name的会话
# 当screen中运行的程序卡住,无法正常终止会话时可以使用
screen -S screen-name -X quit1.2 安装miniconda
1.2.1下载 Miniconda3 安装脚本
wget https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh1.2.2 运行脚本
chmod +x Miniconda3-latest-Linux-x86_64.sh
bash Miniconda3-latest-Linux-x86_64.sh1.2.3 遵循安装向导
-
查看许可协议:按回车键查看协议内容,直到最后。
-
同意许可:输入
yes并按回车键。 -
指定安装位置:默认安装路径为
~/miniconda3,你可以直接按回车键接受默认路径。 -
初始化 Conda:安装完成后,输入
yes初始化 Conda
1.2.4 验证安装
初始化激活conda
/home/abc/miniconda3/bin/conda initsource ~/.bashrcconda --versionconda 24.5.0),则表示安装成功对于Linux/macOS用户:
- 确认Anaconda安装路径:首先,确定Anaconda安装到了哪个目录。默认情况下,它可能安装在
~/anaconda3或~/miniconda3目录下。 -
编辑bash配置文件:打开你的bash配置文件。对于大多数用户,这可能是
~/.bashrc或~/.bash_profile(取决于你的系统和配置)。你可以使用文本编辑器打开它,例如使用vim或nano:nano ~/.bashrc - 添加conda路径到PATH:在文件的末尾添加以下行,替换
/path/to/anaconda3为你的实际Anaconda安装路径:export PATH="/path/to/anaconda3/bin:$PATH" - 使修改生效:保存并关闭文件后,为了让修改立即生效,运行以下命令:
source ~/.bashrc如果你编辑的是
~/.bash_profile,则运行:source ~/.bash_profile - 验证conda命令:现在,在终端中输入
conda --version,如果看到conda的版本信息,说明问题已经解决。
1.2.5 初始化conda 并禁止自动激活环境
在终端中运行以下命令来初始化 Conda
conda init
source ~/.bashrc禁止自动激活conda环境
conda config --set auto_activate_base false
1.2.6可选配置
配置国内镜像加速
conda config --add channels https://mirrors.ustc.edu.cn/anaconda/pkgs/main/
conda config --add channels https://mirrors.ustc.edu.cn/anaconda/pkgs/free/
conda config --set show_channel_urls yes2 安装mamba
conda install mamba -n base -c conda-forge确认安装
安装成功
3 PICRUSt2安装(通过mamba实现)
参照github官网的安装教程,使用下面的命令安装,github:安装 ·picrust/picrust2 维基
mamba create -n picrust2 -c bioconda -c conda-forge picrust2=2.6.2中间会出现确认 安装的地方
 然后等待安装:
然后等待安装:

运行以下命令来初始化当前的 Bash shell,使mamba可以用,不然下面的命令会报错
eval "$(mamba shell hook --shell bash)"需要激活 picrust2环境:
mamba activate picrust2或者使用conda命令来激活创建的picrust2环境(mamba和conda二选一激活虚拟环境即可)
comda activate picrust2现在就已经进入picrust的环境了

4 使用示例数据运行程序
下载并解压缩文件:
wget http://kronos.pharmacology.dal.ca/public_files/picrust/picrust2_tutorial_files/chemerin_16S_arc.tar.gz
tar -xvf chemerin_16S_arc.tar.gz运行 PICRUSt2:
picrust2_pipeline.py -s chemerin_16S_arc/seqs_arc.fna -i chemerin_16S_arc/table.biom -o picrust2_out_pipeline -p 15 --verbose
运行完成,我使用的是15线程,示例数据跑了478s,大约就是8min

查看一下输出的文件目录:
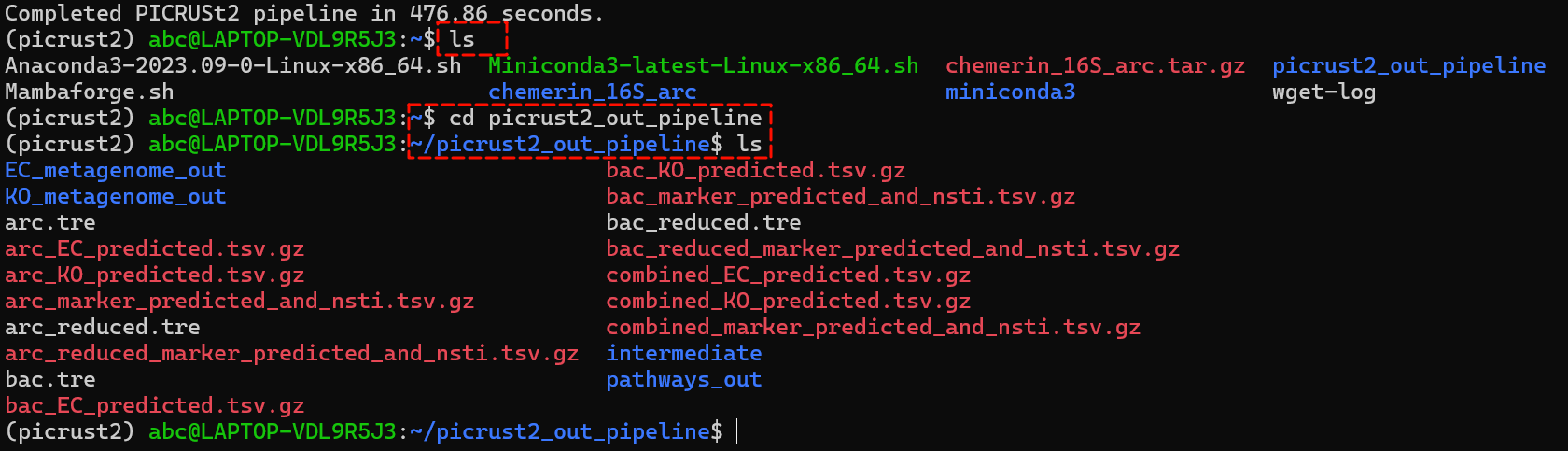
5 添加注释
添加注释信息
添加注释的前提是需要下载add_descriptions.py脚本,地址参考刘永鑫老师的github:picrust2/scripts/add_descriptions.py at master · picrust/picrust2
此外我还将add_descriptions.py上传到网盘上可以直接下载使用,需要将下载好的脚本放到Linux虚拟子系统中,我存放的路径是/home/abc,abc是自己创建的用户名,我使用的是Xftp上传的,参考Windows开启(WSL)Linux子系统并远程连接SSH - leo朵 ,在脚本所在的目录运行下面的命令,或使用脚本的绝对路径也可以执行(注意:要在创建的picrust环境中运行)
# add_descriptions.py添加EC、KO、MetaCyc的注释信息
add_descriptions.py -i picrust2_out_pipeline/KO_metagenome_out/pred_metagenome_unstrat.tsv.gz -m KO -o picrust2_out_pipeline/KO_metagenome_out/pred_metagenome_unstrat_descrip.tsv.gz
add_descriptions.py -i picrust2_out_pipeline/EC_metagenome_out/pred_metagenome_unstrat.tsv.gz -m EC -o picrust2_out_pipeline/EC_metagenome_out/pred_metagenome_unstrat_descrip.tsv.gz
add_descriptions.py -i picrust2_out_pipeline/pathways_out/path_abun_unstrat.tsv.gz -m METACYC -o picrust2_out_pipeline/pathways_out/path_abun_unstrat_descrip.tsv.gz注释后的KO表格如下:
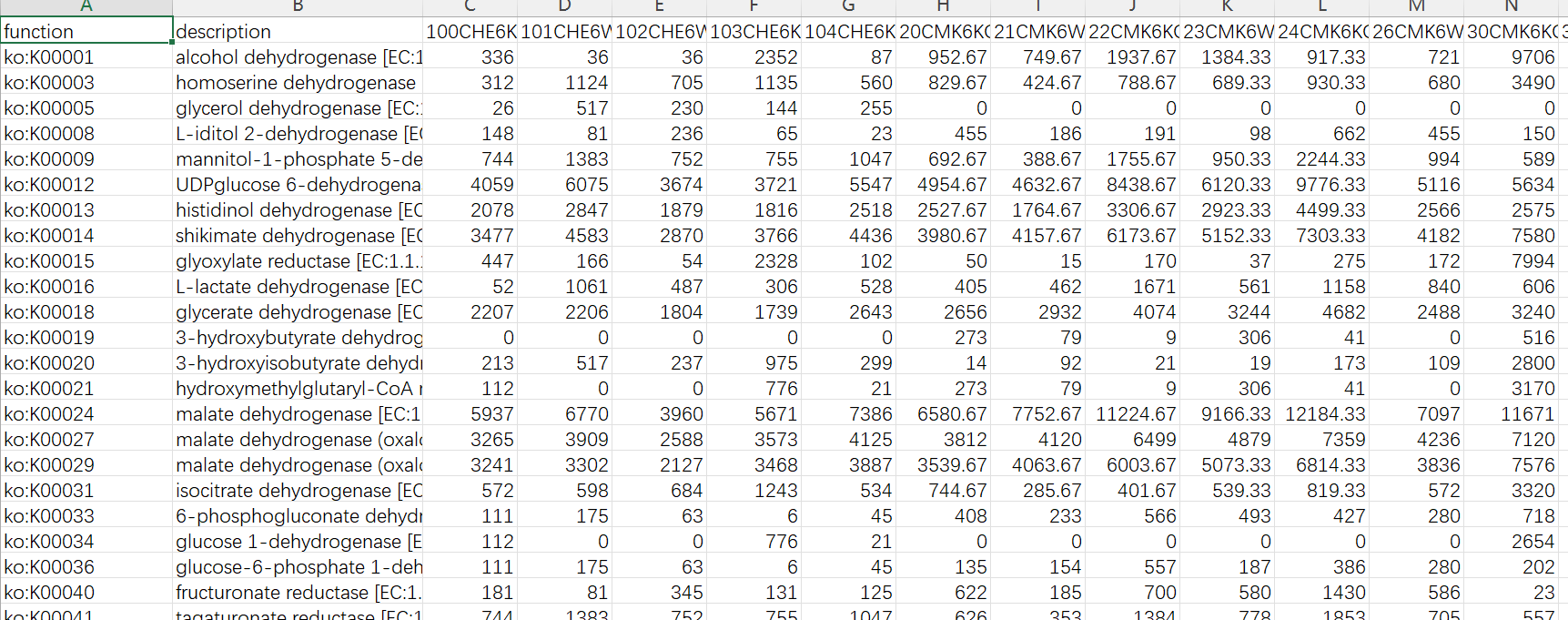
给ko添加KEGG层级信息
注释之前需要下载一个python脚本summarizeAbundance.py和一个txt注释文件KO1-4.txt ,参考地址: EasyMicrobiome/script/summarizeAbundance.py at master · YongxinLiu/EasyMicrobiome,需要将py脚本和KO1-4.txt 文件放在linux虚拟机中,我存放的路径是/home/abc,abc是自己创建的用户名
# 添加ko的通路信息
zcat picrust2_out_pipeline/KO_metagenome_out/pred_metagenome_unstrat.tsv.gz > KEGG.KO.txt
python3 summarizeAbundance.py \
-i KEGG.KO.txt \
-m KO1-4.txt \
-c 2,3,4 -s ',+,+,' -n raw \
-o KEGG
参考资料:
脚本下载
2025-06-18如有失效,请留言